Podcast
Questions and Answers
Quale approccio è stato determinante per la riorganizzazione dell'albero della vita degli eucarioti (eToL)?
Quale approccio è stato determinante per la riorganizzazione dell'albero della vita degli eucarioti (eToL)?
- Confronto dei cicli vitali di piante, animali e funghi.
- Studio approfondito delle interazioni ecologiche tra eucarioti.
- Analisi morfologica dettagliata delle cellule eucariotiche.
- Applicazione diffusa della filogenomica e scoperta di nuovi lignaggi di protisti eterotrofi. (correct)
I supergruppi eucariotici attualmente riconosciuti sono ampiamente supportati da caratteristiche morfologiche e biologiche condivise.
I supergruppi eucariotici attualmente riconosciuti sono ampiamente supportati da caratteristiche morfologiche e biologiche condivise.
False (B)
Qual è stato uno dei principali metodi utilizzati per isolare e studiare nuovi lignaggi di protisti, contribuendo alla revisione dell'eToL?
Qual è stato uno dei principali metodi utilizzati per isolare e studiare nuovi lignaggi di protisti, contribuendo alla revisione dell'eToL?
Colture tradizionali
L'acronimo TSAR, usato per uno dei supergruppi eucariotici, sta per ______, Stramenopili, Alveolati e Rhizaria.
L'acronimo TSAR, usato per uno dei supergruppi eucariotici, sta per ______, Stramenopili, Alveolati e Rhizaria.
Abbina i seguenti supergruppi originali con le loro caratteristiche principali:
Abbina i seguenti supergruppi originali con le loro caratteristiche principali:
Quale delle seguenti affermazioni descrive meglio l'evoluzione della costruzione dell'eToL?
Quale delle seguenti affermazioni descrive meglio l'evoluzione della costruzione dell'eToL?
Il modello dei supergruppi semplifica la comprensione della filogenesi eucariotica evidenziando caratteristiche biologiche condivise tra i gruppi.
Il modello dei supergruppi semplifica la comprensione della filogenesi eucariotica evidenziando caratteristiche biologiche condivise tra i gruppi.
Qual è il significato dell'eToL nello studio della biologia degli eucarioti?
Qual è il significato dell'eToL nello studio della biologia degli eucarioti?
Il supergruppo ______ è caratterizzato dalla presenza di plastidi primari e include alghe verdi, alghe rosse e glaucofite.
Il supergruppo ______ è caratterizzato dalla presenza di plastidi primari e include alghe verdi, alghe rosse e glaucofite.
Abbina i seguenti taxa con la loro descrizione:
Abbina i seguenti taxa con la loro descrizione:
Quale fattore ha contribuito in modo significativo all'aumento della densità di taxa nei dataset filogenomici, come indicato nel testo?
Quale fattore ha contribuito in modo significativo all'aumento della densità di taxa nei dataset filogenomici, come indicato nel testo?
TSAR è una delle suddivisioni degli eucarioti più supportate con un'ampia evidenza di apomorfie.
TSAR è una delle suddivisioni degli eucarioti più supportate con un'ampia evidenza di apomorfie.
Che cosa significa il termine filogenomica?
Che cosa significa il termine filogenomica?
Oltre a TSAR, ______ rappresenta un nuovo supergruppo costituito da collodictyonids (sin. diphylleids) + Rigifilida + Mantamonas.
Oltre a TSAR, ______ rappresenta un nuovo supergruppo costituito da collodictyonids (sin. diphylleids) + Rigifilida + Mantamonas.
Abbina i seguenti gruppi con la loro descrizione:
Abbina i seguenti gruppi con la loro descrizione:
Flashcards
Cos'è l'eToL?
Cos'è l'eToL?
L'albero della vita degli eucarioti rappresenta la filogenesi di tutti i lignaggi eucariotici, comprendendo principalmente 'protisti' microbici.
Cos'è la filogenomica?
Cos'è la filogenomica?
Phylogenomics combina dati su scala genomica con metodi filogenetici, stimando la filogenesi da allineamenti genici di sequenze amminoacidiche.
Cosa significa TSAR?
Cosa significa TSAR?
TSAR è un acronimo per Telonemidi, Stramenopili, Alveolati e Rhizaria, un clade emerso nell'era filogenomica, comprendente metà delle specie eucariotiche.
Cosa sono i CRuMs?
Cosa sono i CRuMs?
Signup and view all the flashcards
Cosa include Discoba?
Cosa include Discoba?
Signup and view all the flashcards
Chi sono gli Hemimastigophora?
Chi sono gli Hemimastigophora?
Signup and view all the flashcards
Cosa comprende Haptista?
Cosa comprende Haptista?
Signup and view all the flashcards
Cosa contiene Cryptista?
Cosa contiene Cryptista?
Signup and view all the flashcards
Cosa comprende Archaeplastida?
Cosa comprende Archaeplastida?
Signup and view all the flashcards
Cosa raggruppa Amorphea?
Cosa raggruppa Amorphea?
Signup and view all the flashcards
A cosa porta la scoperta di nuovi protisti?
A cosa porta la scoperta di nuovi protisti?
Signup and view all the flashcards
Perché il modello dei supergruppi è astratto?
Perché il modello dei supergruppi è astratto?
Signup and view all the flashcards
Quali studi aggiungono nuovi lignaggi?
Quali studi aggiungono nuovi lignaggi?
Signup and view all the flashcards
Quali gruppi compongono TSAR?
Quali gruppi compongono TSAR?
Signup and view all the flashcards
Quali organismi formano CRuMs?
Quali organismi formano CRuMs?
Signup and view all the flashcards
Study Notes
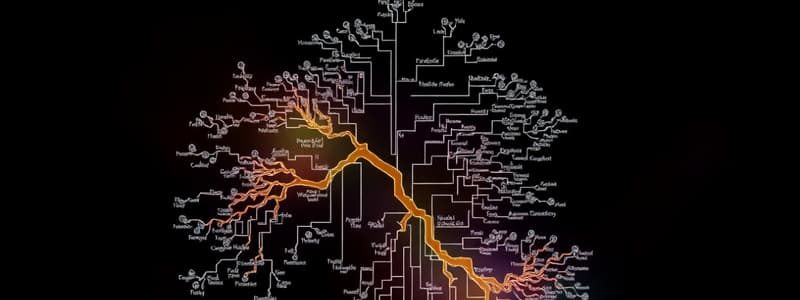
La Nuova Albero degli Eucarioti
- Per 15 anni, l'Albero della Vita degli eucarioti (eToL) è stato suddiviso in cinque-otto raggruppamenti principali, noti come "supergruppi".
- L'Albero della vita degli eucarioti è stato profondamente riorganizzato in questo periodo.
- Il nuovo eToL deriva dall'applicazione diffusa della filogenomica e da numerose scoperte di importanti lignaggi di eucarioti, per lo più protisti eterotrofi a vita libera.
- Le prove a sostegno dell'Albero della vita degli eucarioti sono passate da una sintesi di filogenetica molecolare e caratteri biologici alla pura filogenetica molecolare.
- La maggior parte degli attuali supergruppi manca di caratteristiche morfologiche o cellulari-biologiche che li definiscano, rendendo l'etichetta di supergruppo ancora più arbitraria di prima.
- In futuro, la combinazione di colture tradizionali con approcci senza coltura in via di maturazione e la filogenomica dovrebbe accelerare il processo di completamento e risoluzione dell'eToL ai suoi livelli più profondi.
L'Albero della Volta degli eucarioti
- Risolvere l'albero evolutivo per tutti gli eucarioti è stato un obiettivo a lungo termine della biologia.
- Inferire un eToL che sia accurato e completo è un obiettivo valido di per sé, ma l'eToL è anche la struttura su cui comprendiamo le origini e la storia della biologia degli eucarioti e dei processi evolutivi che la sostengono.
- L'eToL è quindi uno strumento fondamentale per studiare molti aspetti dell'evoluzione degli eucarioti, come la biologia cellulare, l'organizzazione del genoma, il sesso e la multicelluarità.
- Nell'era molecolare, l'eToL è diventato anche una risorsa fondamentale per interpretare i dati di sequenza ambientale e quindi rivelare la diversità e la composizione delle comunità ecologiche.
- La maggior parte delle specie descritte di eucarioti appartiene ai gruppi multicellulari di animali (metazoi), piante terrestri e funghi (3 regni).
- È chiaro da tempo che questi tre "regni" rappresentano solo una piccola proporzione della diversità eucariotica di alto livello.
- La stragrande maggioranza di questa diversità è rappresentata dai "protisti", gli eucarioti che non sono animali, piante o funghi.
- Risolvere l'eToL significa risolvere le relazioni tra i principali lignaggi di protisti. Il compito è complicato dal fatto che i protisti sono molto meno studiati nel complesso rispetto agli animali, alle piante o ai funghi.
- I dati di sequenza molecolare si sono accumulati lentamente per molti taxa di protisti noti e numerosi lignaggi importanti erano completamente sconosciuti (o non erano coltivati, quindi difficili da studiare) quando è iniziata l'era molecolare.
- Risolvere l'eToL è stato un processo in cui la scoperta su larga scala di lignaggi principali è avvenuta contemporaneamente all'inferenza filogenetica di livello profondo.
Il modello per Supergruppi
- Nei primi anni 2000, è emerso un modello dell'albero che divideva quasi tutta la diversità eucariotica conosciuta tra cinque-otto taxa principali, di solito indicati come "supergruppi".
- La categoria di supergruppo era puramente informale, a indicare raggruppamenti estremamente ampi che comprendevano, ad esempio, i tradizionali "regni" come Metazoi e Funghi come sottocladi.
- I supergruppi originali rappresentavano generalmente le collezioni più inclusive di organismi all'interno degli eucarioti per i quali esistevano prove ragionevoli che formassero un gruppo monofiletico.
- Un elenco tipico di questi gruppi comprendeva (con alcune differenze nella capitalizzazione e nelle terminazioni): Archaeplastida (noti anche come Plantae), Chromalveolata, Rhizaria (o Cercozoa), Opisthokonta, Amoebozoa ed Excavata.
- Le principali variazioni tra i resoconti di quel tempo erano che alcuni univano Opisthokonta ed Amoebozoa come "unikonts" (poi rinominati "Amorphea") o non mostravano Excavata e/o Chromalveolata risolti con sicurezza come cladi.
- Per la metà dei gruppi (cioè Opisthokonta, Amoebozoa e Rhizaria), la prova principale a sostegno della loro unità era la filogenesi di uno o pochi geni.
- Per gli altri, era una combinazione di prove filogenetiche molecolari più deboli e caratteristiche cellulari-biologiche derivate condivise.
- Archaeplastida e Chromalveolata sono stati ciascuno identificati dalla presenza di plastidi simili, con sequenze di genomi plastidici che supportano un'origine endosimbiotica ancestrale dei plastidi in ciascun gruppo.
- Gli Excavata, nel frattempo, si distinguevano per l'inferenza che i taxa condividessero un apparato citoscheletrico flagellare derivato e complesso.
- Gli eToL originali basati sui supergruppi erano sintesi di informazioni diverse piuttosto che semplici sintesi delle filogenesi molecolari.
- Il modello dei supergruppi per l'eToL è diventato ampiamente popolare sia nella letteratura primaria che nei libri di testo per diversi motivi.
- Il modello ha reso possibile la sintesi degli eucarioti in modo conveniente ed efficiente, poiché quasi tutte le specie rientravano in questi pochi gruppi principali relativamente diversi.
- Tutti i supergruppi originali, tranne i Rhizaria, avevano almeno una caratteristica biologica distintiva che sembrava definirli ancestralmente Box 1.
- I raggruppamenti sembravano coincidere con i limiti della risoluzione filogenetica.
- Il modello di supergruppo globale è rimasto la descrizione standard dell'eToL per 15 anni, nonostante i principali cambiamenti nella nostra conoscenza della filogenesi e della diversità eucariotica nel corso del tempo.
Nuovi Metodi e Taxa
- I profondi cambiamenti all'eToL sono derivati dallo sviluppo della filogenomica e, contemporaneamente, dall'aggiunta di molti lignaggi di protisti evolutivamente importanti ai set di dati molecolari.
Filogenomica
- Il termine "filogenomica" copre vari approcci che combinano dati su scala genomica con metodi filogenetici.
- Nel contesto dell'eToL, di solito si riferisce alla stima della filogenesi organismica da set di dati contenenti dozzine o centinaia di allineamenti genici, il più delle volte geni codificati nel nucleo analizzati come sequenze di amminoacidi inferti.
- I dati provengono da un mix di progetti di sequenziamento del genoma e, frequentemente, del trascrittoma.
- L'introduzione della filogenomica ha offerto la promessa di superare le informazioni limitate fornite da singoli geni, che per lo più erano inadeguate a risolvere profonde divergenze all'interno dell'eToL.
- I fenomeni che causano il raggruppamento di taxa non correlati nelle filogenesi, come il bias compositivo e gli alti tassi di divergenza della sequenza, spesso influenzano anche l'intero genoma
- Pertanto, la semplice aggiunta di geni può amplificare gli artefatti piuttosto che annullarli.
- L'accuratezza potrebbe essere migliorata utilizzando modelli evolutivi più realistici e, soprattutto, una scelta accurata dei taxa, ove possibile.
- L'esame di più geni solleva anche lo spettro di combinare artificialmente storie genetiche diverse, rendendo essenziali controlli di qualità accurati per eliminare assegnazioni di paralogo errate, sequenze contaminanti, ecc.
- Durante il sequenziamento (denominato contaminazione sul sequenziatore/cella di flusso), durante la preparazione della libreria o nella coltura cellulare può verificarsi contaminazione.
Nuovi e Riscoperti Taxa Principali
- Quando è emerso il modello di supergruppo, sembrava possibile che la maggior parte dei lignaggi principali fosse già stata scoperta, sulla base di diverse linee di evidenza.
- Molti degli ex "eucarioti misteriosi" che erano stati esaminati utilizzando strumenti molecolari (cioè, era nota almeno una sequenza di rDNA 18S) erano stati assegnati a gruppi principali esistenti; ad esempio, molti piccoli flagellati e amebe rientravano nei Rhizaria
- C'era solo un piccolo elenco di protisti non sequenziati che erano buoni candidati a rappresentare "nuovi" lignaggi principali perché era noto che avevano una struttura cellulare insolita.
- Accurate analisi di dati molecolari ambientali avevano dimostrato che quasi tutte le sequenze di rRNA eucariotiche disponibili potevano essere assegnate a gruppi principali noti.
- Dal 2004, tuttavia, è stato scoperto un numero notevole di nuovi protisti che potrebbero essere cruciali per la comprensione dell'eToL ai livelli più profondi e una serie di ri-isolamenti di taxa noti ma non sequenziati che si sono dimostrati altrettanto importanti.
- Quasi tutti questi sono flagellati o amebe eterotrofi a vita libera.
- La grande maggioranza è stata isolata e resa disponibile per lo studio molecolare utilizzando approcci di coltura "vecchio stile" piuttosto che i metodi molecolari ambientali ad alta produttività che altrimenti hanno trasformato la ricerca sulla diversità dei protisti.
- La maggior parte di questi nuovi lignaggi di protisti sono rari o addirittura non rilevati nei dati molecolari ambientali provenienti da sistemi ben studiati come il pelagico marino e/o includono solo un piccolo numero di specie conosciute (spesso solo una o due).
- L'aggiunta di questi taxa alle analisi filogenomiche, soprattutto negli ultimi 5 anni, ha trasformato il catalogo dei rami che compongono la struttura di livello profondo dell'eToL.
L'Albero Corrente
- Integrando i risultati delle analisi filogenomiche e i principali lignaggi aggiunti negli ultimi 15 anni, l'albero di consenso attuale è stato modificato al punto che la maggior parte dei supergruppi originali sono stati sussunti in nuovi taxa o sono scomparsi del tutto.
- Le modifiche sono derivate da tre processi principali: la suddivisione di vecchi supergruppi, spesso seguita dalla riorganizzazione in diverse parti dell'albero; l'amalgamazione di taxa isolati in nuovi cladi più grandi; e l'aggiunta all'ingrosso di nuovi taxa a livello di supergruppo.
- Il risultato è un modello dell'eTol che ha mantenuto una forma complessiva simile a quella dei precedenti schemi ampi, ma i cui dettagli sono molto diversi.
TSAR
- L'acronimo TSAR sta per i membri costituenti del gruppo: telonemidi, stramenopili, alveolati e Rhizaria.
- Gli ultimi tre gruppi formano un clade, "SAR" o "Sar", emerso relativamente presto nell'era filogenomica ed è stato considerato di routine un "supergruppo" (in parte sostituendo i cromalveolati).
- Si stima che SAR comprenda fino alla metà di tutte le specie di eucarioti.
- Il gruppo fratello di Sar non era chiaro, ma ora ci sono buone prove che questo sia l'enigmatico taxon di flagellati a vita libera Telonemia, che ha solo due specie descritte.
- Il clade TSAR è stato robustamente supportato in recenti analisi filogenomiche con una migliore qualità e quantità di sequenze ed è stato anche recuperato in precedenza con alcuni set di dati più piccoli.
Haptista
- Haptista comprende le alghe aptofite (precedentemente assegnate ai cromalveolati e centroelidi.
- Gli aptofiti, in particolare i coccolitoforidi calcificanti, svolgono un ruolo cruciale negli ecosistemi marini e nei cicli biogeochimici globali.
- I centroelidi, al contrario, sono protozoi a vita libera con pseudopodi simili a raggi supportati da microtubuli (axopodia), che si irradiano da un corpo cellulare sferico.
- Haptista è generalmente ben supportato in recenti studi filogenomici.
Cryptista
- Cryptista contiene i criptomonadi (anche ex cromalveolati; un lignaggio che è stato centrale nello studio dell'origine e della diffusione dei plastidi attraverso gli eucarioti.
- Cryptista comprende anche i katablepharidi e i più recentemente scoperti Palpitomonas, entrambi enigmatici flagellati eterotrofi.
- Gli studi filogenomici supportano robustamente la monofilia di Cryptista.
Archaeplastida
- I tre taxa che compongono Archaeplastida sono Chloroplastida (alghe verdi + piante terrestri), Rhodophyta (alghe rosse) e Glaucophyta.
- Tutti e tre i lignaggi hanno plastidi primari, che sono organelli fotosintetici che hanno avuto origine direttamente dai cianobatteri.
- Recentemente, un nuovo gruppo - Rhodelphis - è stato scoperto e dimostrato di ramificarsi come sorella delle alghe rosse nelle analisi filogenomiche.
- Le cellule di rodelphis sono flagellati eterotrofi, ma i dati di sequenza genetica suggeriscono che hanno un plastidio primario non fotosintetico.
- L'ipotesi di un'origine comune dei plastidi primari unifica gli Archaeplastida e, unicamente tra gli attuali supergruppi, implica una forte sinapomorfia morfologica/cellullare-biologica.
- La maggior parte delle recenti analisi filogenomiche dei geni nucleari non recupera gli Archaeplastida come un clade stretto o lo fa con scarso supporto.
- Le topologie alternative più comuni collocano Cryptista e talvolta Picozoa (vedi sotto) all'interno del clade minimo verde + rosso + glaucofite.
Amorphea
- Questo taxon raggruppa gli opistoconti (animali, funghi e relativi parenti unicellulari) con i protisti ameboidi di Amoebozoa.
- Amorfia ora include anche due piccoli lignaggi di flagellati eterotrofi, i breviati e gli apusomonadi, che si raggruppano con gli opistoconti per formare l'Obazoa.
- Amorphea è robustamente supportato nella maggior parte delle analisi filogenomiche, con l'avvertenza che la posizione della radice rimane incerta e un posizionamento all'interno di Amorphea è stato dedotto in alcuni casi, il che renderebbe Amorphea parafiletico.
CRuMs
- Come con TSAR, CRuMs rappresenta un nuovo supergruppo proposto chiamato come acronimo dei suoi membri costituenti: collodictyonidi (sin. difilleidi) + Rigifilida + Mantamonas.
- Questi tre taxa di protozoi a vita libera hanno morfologie di base molto diverse (flagellati natatori, cellule ameboidi filose e piccole cellule plananti, rispettivamente) ed erano precedentemente "taxa orfani" ma si sono uniti robustamente a seguito di recenti analisi filogenomiche.
Discoba
- Discoba comprende Euglenozoa ed Heterolobosea (collettivamente 'Discicristata'), più i gruppi di flagellati eterotrofi Jakobida e Tsukubamonas.
- Euglenozoa comprende le alghe euglenofite, i parassiti tripanosomatidi e numerosi flagellati eterotrofi a vita libera o parassiti, che sono abbondanti in molti ecosistemi.
- Gli eterolobosei sono amebe e flagellati eterotrofi.
- Discoba era sospettato sulla base di filogenesi a singolo e multigene selezionate e fortemente confermato da analisi filogenomiche.
Metamonada
- I metamonada comprendono interamente protisti anaerobici, tra cui vari protozoi a vita libera, simbionti intestinali (specialmente di insetti mangiatori di legno) e molti parassiti.
- La monofilia di Metamonada è ben supportata dalle analisi filogenomiche contemporanee.
- Collocare i metamonada rispetto ad altri taxa si è rivelato molto impegnativo, perché la maggior parte delle specie presenta tassi di evoluzione della sequenza molto elevati.
- Le analisi filogenomiche spesso deducono un clade Metamonada più Discoba (vedi sopra) corrispondente ampiamente al supergruppo "Excavata" originale; tuttavia, questa topologia potrebbe rappresentare un artefatto di analisi.
- Alcune analisi filogenomiche, di solito quelle che includono metamonada a ramificazione più corta, recuperano invece una relazione specifica con il gruppo "orfano" di escavati malawimonadi.
Hemimastigophora
- Gli "emimastigoti" sono protozoi a vita libera con due file di flagelli.
- Sono noti fin dal XIX secolo e hanno ricevuto un alto rango tassonomico sulla base di osservazioni di microscopia elettronica, ma non sono mai stati coltivati e mancavano dati genetici.
- Le recenti analisi filogenomiche, basate su trascrittomi da celle selezionate manualmente di due generi, hanno mostrato gli emimastigoti come uno dei rami più profondi all'interno degli eucarioti.
- Non potevano essere collocati come sorella di nessuno dei supergruppi "stabiliti" (o "orfani"); di conseguenza, è stato proposto di considerarli un nuovo supergruppo.
- Diaphoretickes" è un assemblaggio ancora più grande che è stato proposto per unire questi quattro supergruppi escludendo Amorphea, Discoba e Metamonada, mentre CRuMs si deduce che sia sorella di Amorphea.
- È troppo presto per dirlo, ma anche se affidabili, queste inferenze dipendono da ipotesi sulla posizione della radice degli eucarioti, che diventa sempre più problematica man mano che gruppi più grandi vengono dedotti da alberi filogenetici non radicati.
Taxa Orfani
- Oltre ai gruppi sopra elencati, ci sono diversi taxa apparentemente poveri di specie per i quali le analisi filogenomiche finora non sono riuscite a fornire un posizionamento filogenetico convincente.
- Questi cosiddetti "taxa orfani" includono Ancoracysta, Picozoa, malawimonads e ancyromonads (= planomonads), tutti protozoi a vita libera.
- Alcuni o tutti questi potrebbero ramificarsi con un gruppo stabilito; ad esempio, Ancoracysta può essere sorella di Haptista e i malawimonadi possono essere sorella di Metamonada.
- È possibile, tuttavia, che alcuni rappresentino lignaggi divergente anche più profondi, seguendo il recente esempio di Hemimastigophora.
La Natura del Modello di Supergruppo
- Oltre all'elenco dei supergruppi che cambia notevolmente negli ultimi 15 anni, è cambiata anche la natura tipica di tali raggruppamenti, con importanti conseguenze sul modo in cui concettualizziamo il modello di supergruppo per descrivere l'albero.
- Come accennato in precedenza, la maggior parte dei supergruppi originali si distingueva per qualche caratteristica biologica evidente.
- Al contrario, i nuovi supergruppi riflettono principalmente cladi in alberi filogenetici privi di caratteristiche derivate condivise candidate.
- Oggi, tuttavia, l'ipotesi cromalveolata non è ampiamente accettata (anche se vedi per un'opinione diversa), principalmente perché la maggior parte delle analisi filogenetiche non mostra strette relazioni tra i lignaggi ospiti.
- Nessuno dei nuovi raggruppamenti derivanti dalla disintegrazione dei cromalveolati (TSAR, Cryptista, Haptista) è probabilmente definito ancestralmente dai plastidi secondari rossi.Una simile osservazione può essere fatta per Opisthokonta, che rimane distinto per avere ancestralmente un singolo flagello posteriore nelle cellule mobili, ma ora è solitamente considerato un sottotassone all'interno di Obazoa e Amorphea, nessuno dei quali ha caratteristiche morfologiche unificanti.Anche i supergruppi che si distinguono ancora per una proprietà biologica sono tra i più instabili, almeno con le loro attuali composizioni. Archaeplastida è definito dalla presenza di plastidi primari, ma rimane scarsamente supportato dalle analisi filogeneticheA allo stesso modo, Metamonada consiste interamente di anaerobi, ma è probabile sia sussunto in un taxon una volta che le relazioni tra gli 'escavati' siano meglio comprese.Le decisioni su quali siano i principali raggruppamenti considerati supergruppi sono sempre state arbitrarie, ma la crescente assenza di distinte caratteristiche biologiche lo rende ancora più evidente. Paradossalmente, una migliorata risoluzione dell'albero peggiora il problema, non migliorarlo.Per illustrare questo problema, si prenda la supergruppo di nuova identificazione CRuMS, che si deduce si ramifica insieme all'Amorfea, stessa contenente due taxa spesso riconosciuti come supergruppi di supergruppi, Amoebozoa e Obazoa.L'opinione che questa collezione di taxa in rappresentanza due supergruppi (o tre) piuttosto che uno, riflette la mancanza di caratteri distintivi per il raggruppamento crums-amorphea.Questo lascia guidata la decisione dal giudizio soggettivo riguardante: e che risultati filogenetici sono sufficientemente robusti da essere accettati senza ulteriore conferma; e ii l 'incertezza circa l'ubicazione della "radice" di etol.Inoltre, c'è un una linea sfocata tra lignaggi orfani, che spesso hanno solo poche specie note e i supergruppi meno speculativi.se un orfano povero di diversità dimostra di essere evolutivamente estraneo a tutti i supergruppi, questo lo rende un nuovo supergruppo?per essere più utili, la nozione di 'supergruppo' non dovrebbe essere distinta da 'orfano' dal livello di versatilità che contiene, ma invece dovrebbe riflettere il grado di fiducia che un lignaggio non è racchiuso filogeneticamente da un clade esistente.Ci aspettiamo che molti ricercatori e gli educatori continueranno a trovare utile dividere la diversità eucariotica in un piccolo numero di Clade principali e questo alla fine è ciò che un catalogo di supergruppi ha lo scopo di fornire la ricerca genonica comparativa potrebbe identificare apomorfia robusti per il Deep Clade all'interno di Eukaryotes, che a turno potrebbe aiutare più naturalmente a delineare i supergruppi fino ad allora, tuttavia, sembra che la maggior parte delle maggiori suddivisioni di Eucarioti continuerà ad essere Clade derivato solo dagli alberi filogenetici molecolari di conseguenza, dovremmo aspettarci che l'elenco dei supergruppi da sempre più volate man mano che la comprensione della diversità degli eucarioti e risoluzione dell'albero migliora ulteriormente e più autore dipendente, poiché non ci saranno criteri evidenti con cui decidere quali Clade devono essere distinte come supergruppi.
Dove Stiamo Andendo?
- Le recenti scoperte di diversi rami molto profondi neli' etol hanno consentito una profonda rivalutazione delle principali transizioni evolutive avvenute centinaia di milioni di anni fa.
- Sorprendentemente, questi organismi sono stati per lo più identificati utilizzando approcci culturali classici a bassa produttività e il tasso di tali scoperte non mostra segni di calo.
- In parallelo, tuttavia, stiamo assistendo a una rapida maturazione e a un grande aumento dell'accessibilità di metodi trascrittomici e genomici a cella singola, che non si basano sulla coltivazione.
- Un gran numero di cellule può essere isolato in blocco dall'ambiente, quindi sottoposto a screening utilizzando tecniche molecolari per identificare organismi importanti per ulteriori studi; in alternativa, le cellule bersaglio possono essere identificate mediante microscopia e selezionate individualmente.
- Sia la genomica a cella singola che la trascrittomica utilizzano tecniche di amplificazione e in genere generano assemblaggi relativamente incompleti e rappresentazioni distorte.
- Tuttavia, l'approccio trascrittomico, almeno per le cellule più grandi, può produrre un'ottima copertura nei set di dati filogenomici, che sono dominati da geni di mantenimento ad alta espressione.
- È inoltre necessario un ulteriore sviluppo di metodi sistematici a più alta produttività per esplorare la frazione eucariotica microbica degli ecosistemi.
- La metagenomica (o metatrascrittomica) ha rivoluzionato la ricerca sui procarioti, fornendo migliaia di genomi ricostruiti per organismi che potrebbero non essere mai stati osservati al microscopio.
- La metagenomica eucariotica è ancora agli inizi, ma ci sono segnali che potrebbe essere un approccio praticabile per collocare una nuova diversità genetica in una struttura filogenomica.
- Un altro metodo recentemente applicato per ottenere informazioni genomiche da taxa importanti combina il metabarcoding e l'ibridazione fluorescente in situ (FISH) per tornare dalle sequenze alle cellule.
- Non importa quale tecnica si dimostri la più utile, la liberazione dall'onere della coltivazione significa che l'ampiezza tassonomica e, soprattutto, la densità tassonomica, dei set di dati filogenomici possono migliorare rapidamente nel prossimo futuro.
- In questo modo, nuovi gruppi particolarmente difficili da coltivare possono essere identificati e aggiunti per la prima volta, a loro volta accelerando notevolmente il raggiungimento di un robusto campionamento dei taxa per tutti i gruppi sull'albero.
- La disponibilità di questi dati dovrebbe in definitiva migliorare l'affidabilità complessiva della stima filogenetica, anche se con l'avvertenza che l'utilizzo di approcci senza coltura in genere significa che alcuni aspetti importanti della biologia vengono persi (ad esempio, dettagli dei cicli di vita e morfologia ).
- Con questi miglioramenti previsti nel campionamento dei taxa per gli eucarioti, è più importante che mai sviluppare solide pipeline filogenomiche.
- Ciò comporta le migliori pratiche durante l'assemblaggio dei set di dati e modelli di evoluzione della sequenza abbastanza complessi da descrivere adeguatamente i processi in atto, con implementazioni software che consentono l'utilizzo di questi modelli su set di dati di grandi dimensioni.
- Finora, la filogenomica su vasta scala degli eucarioti ha quasi esclusivamente utilizzato l'approccio di concatenazione, ma l'esplorazione, in profondità, dell'influenza dei singoli geni può aiutare a individuare più specificamente dove e come è distribuito il segnale filogenetico.
- Sarà anche utile valutare le origini dei diversi segnali tra diversi set di dati in modo che l'influenza del campionamento di taxa e genici possa essere districata.
- Una migliore comprensione dei set di dati filogenomici a livello eucariotico, combinata con i miglioramenti nei metodi filogenetici all'avanguardia, consentirà il recupero di segnali filogenetici ancora più antichi e difficili da discernere.
Conclusione
- L'eToL è stato notevolmente rimodellato negli ultimi 15 anni a seguito dello sviluppo della filogenomica e dell'aggiunta di taxon di protisti evolutivamente chiave.
- Il supporto per i gruppi principali è passato dall'essere una sintesi di varie prove filogenetiche molecolari e caratteri biologici all'essere basato quasi interamente su filogenesi molecolari multigene.
- Una conseguenza indiretta ma importante di questo cambiamento è che è sempre più difficile descrivere l'albero in termini semplici, anche se la risoluzione dell'albero stesso è notevolmente migliorata e continua a farlo.
- L'eToL sarà sempre un "lavoro in corso" e con le variazioni incrementali nel tempo la nostra comprensione delle relazioni evolutive avanza.
- Con la maturazione dell'approccio filogenomico, siamo meglio attrezzati che mai sia per migliorare la risoluzione dell'eToL sia per facilitarne l'interpretazione alla luce della quantità senza precedenti di dati generati per una crescente diversità di protisti.
Studying That Suits You
Use AI to generate personalized quizzes and flashcards to suit your learning preferences.